Duplication (génétique)

En génétique, la duplication génétique correspond à la multiplication de matériel génétique sur un chromosome. Il existe plusieurs mécanismes qui résultent de la duplication soit d'une large portion chromosomique, soit d'un gène ou bien d'une suite nucléotidique. Ces remaniements du génome représentent un moteur important dans l'évolution des génomes. Le doublement d'un gène crée une copie supplémentaire dégagée de la pression de sélection, ce qui peut permettre à la copie de muer à nouveau sans conséquences nuisibles à l'organisme. C'est un des mécanismes importants de l'évolution moléculaire.
Cependant, dans de nombreux cas, ces altérations sont responsables de maladies génétiques à cause du surplus de données génétiques conduisant à des problèmes au cours du développement. De plus, l'amplification génique peut contribuer à la croissance d'une tumeur. Par exemple l'oncogène C-myc est souvent amplifié dans de nombreuses tumeurs[1],[2].
Duplication du génome
[modifier | modifier le code]Au cours de leur évolution, plusieurs espèces eucaryotes ont subi une duplication complète de leur génome ; on parle alors de paléopolyploïdie. Par exemple, chez la Levure du boulanger (Saccharomyces cerevisiae), le génome s'est dupliqué il y a 100 millions d'années[3].
Mécanismes
[modifier | modifier le code]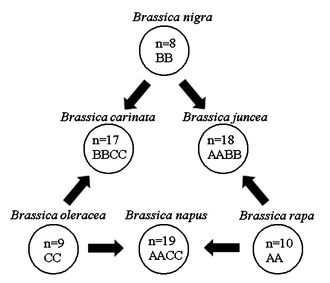
Il existe deux mécanismes permettant la duplication complète d'un génome :
- autopolyploïdie : la non-disjonction des chromosomes dans la lignée germinale au cours de la méiose crée des gamètes diploïdes. La fusion de deux gamètes 2n crée un zygote 4n ;
- allopolyploïdie : l'hybridation interspécifique entre deux espèces résulte en deux ensembles de chromosomes. Chez l'hybride F1, le nombre de chromosomes va doubler et créer une nouvelle espèce si l'hybride est fertile. Par exemple, les plantes du genre Brassica ont évolué par allopolyploïdie. Le triangle d'U est une théorie décrivant les relations génétiques entre les trois espèces Brassica diploïdes (B. oleracea, B. rapa, et B. nigra) et les trois espèces allotétraploïdes (B. napus, B. juncea, et B. carinata) résultantes d'hybridation entre les espèces diploïdes.
Exemples
[modifier | modifier le code]Le génome de nombreuses plantes est polyploïde. On peut citer le blé qui est hexaploïde (6 copies de son génome). En 2007, une collaboration franco-italienne a permis de séquencer le génome de la vigne (Vitis vinifera) : l'ancêtre des plantes dicotylédones a subi plusieurs évènements de duplication de son génome après la divergence des monocotylédones et des dicotylédones ; la plante ancestrale dicotylédone devait être hexaploïde. L'hypothèse émise serait que cette duplication du génome est à l'origine de la radiation des plantes dicotylédones[4].
Le génome des Vertébrés a de même fait l'objet de trois multiplications[5] :
- à la base des Vertébrés[a], une duplication autopolyploïde ;
- à la base des Cyclostomes[b], une triplication autopolyploïde ;
- à la base des Gnathostomes[c], une seconde duplication, allopolyploïde. L'allopolyploïdie entraîne des déséquilibres génomiques qui conduisent à des réarrangements des chromosomes, des changements dans la structure de la chromatine, la méthylation d'une partie de l'ADN et l'activation d'éléments transposables, donc à des innovations et des diversifications : cette seconde duplication pourrait être à l'origine de la réussite évolutive des Gnathostomes[5].
Plasticité des génomes
[modifier | modifier le code]La duplication des génomes est rare car la modification de la dose génique chez un embryon est souvent létale. Par contre, 30 à 80 % des plantes sont polyploïdes et l'explosion du nombre d'espèces chez les angiospermes corrèle avec la duplication complète du génome d'une plante ancestrale. Cela s'explique par une plus grande tolérance au changement du nombre de chromosomes chez les angiospermes par rapport aux animaux[6]. Des duplications du génome récentes datant de moins de 150 ans ont été identifiées chez de nombreuses plantes tels que chez les salsifis où les espèces Tragopogon mirus et Tragopogon miscellus sont apparues il y a environ 80 ans par allotétraploïdie. Ces découvertes permettent de comprendre les conséquences phénotypiques et génétiques du changement de ploïdie[7].
Duplication du génome chez les vertébrés
[modifier | modifier le code]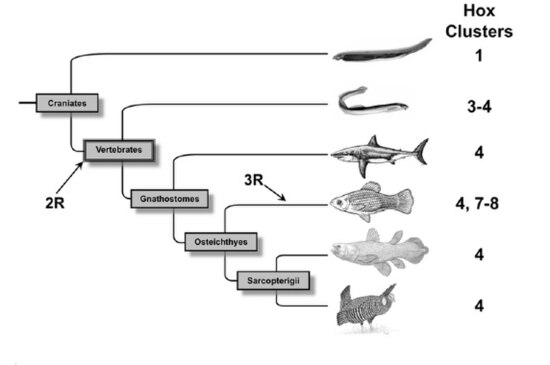
De nombreuses preuves s'accumulent démontrant que pendant l'évolution des vertébrés plusieurs duplications complètes du génome ont eu lieu. Après l'explosion cambrienne il y a environ 450 millions d'années, une première duplication du génome d'un ancêtre des Chordés a eu lieu suivi environ 10 millions d'années plus tard au début de la période Dévonienne d'une deuxième duplication complète du génome. À la fin du Dévonien, une troisième duplication du génome a eu lieu chez les poissons à nageoires rayonnées.
Hypothèse 2R
[modifier | modifier le code]Les premières estimations du nombre de gènes chez l'Homme ont montré que le génome des vertébrés est plus complexe que celui des invertébrés. Pour expliquer l'augmentation de la taille des génomes de vertébrés, Susumu Ohno proposa en 1970 que le génome des vertébrés est le résultat d'une ou plusieurs duplications complètes du génome[8]. Aujourd'hui, cette hypothèse est connue sous le nom d'hypothèse 2R (2 Round) car elle stipule qu'il y a 450 millions d'années une espèce vertébrée ancestrale a subi deux évènements de duplication du génome[9]. Au cours des années 90, cette hypothèse fut extrêmement discutée car les vestiges des duplications géniques disparaissent avec l'évolution. Cependant, aujourd'hui cette hypothèse a recueilli de nombreuses preuves génétiques et phylogénétiques[10]. Un des premiers arguments fut l'absence de liaison entre la plupart des gènes paralogues. En effet, à la suite d'une duplication segmentale les gènes dupliqués sont présents sur le même chromosome et donc génétiquement liés. Par contre, à la suite d'une duplication du génome, les gènes dupliqués se trouvent sur des chromosomes différents. Enfin, l'argument majeur en faveur de l'hypothèse 2R est venu de l'étude des complexes de gènes Hox qui suivent la règle 4:1 c'est-à-dire qu'il existe un complexe Hox chez les invertébrés et quatre complexes Hox chez la plupart des vertébrés qui serait le résultat de deux événements de duplication du génome.
Paralogon Hox
[modifier | modifier le code]
Un complexe Hox est un ensemble de gènes homéotiques groupés sur un chromosome codant des facteurs de transcription essentiels à l'établissement de l'axe antéro-postérieur et l'identité segmentaire chez les animaux. Les quatre complexes Hox chez l'Homme sont sur les chromosomes 2, 7, 12 et 17. À proximité de chaque complexe se trouve un gène paralogue appartenant à la famille Dlx, Collagen et/ou ErbB. Ainsi, le paralogon Hox correspond aux complexes Hox associés aux gènes paralogues situés à proximité du complexe Hox. La présence de plusieurs gènes paralogues à proximité des complexes Hox est un argument majeur de l'hypothèse 2R. Cependant, des analyses phylogénétiques ont montré que certains gènes paralogues et les gènes Hox avaient une histoire évolutive différente ce qui contredit l'hypothèse 2R. Récemment, une étude propose que deux translocations réciproques au sein des complexes Hox intervenues après la deuxième duplication complète du génome expliqueraient les différences d'évolution au sein du paralogon Hox[11].
Hypothèse 3R
[modifier | modifier le code]En 1998, sept complexes Hox ont été identifiés chez le poisson zèbre Danio rerio. Les auteurs de cette étude proposèrent qu'après les deux duplications de génome survenues chez un vertébré ancestral, un troisième évènement de duplication du génome eut lieu spécifiquement chez les poissons téléostéens qui donna huit complexes Hox suivi de la perte d'un complexe Hox[12].
Hypothèse 4R
[modifier | modifier le code]En 2005, une équipe canadienne démontra qu'il existait chez le saumon de l'Atlantique et la truite en arc-en-ciel qui appartiennent à la famille des salmonidés, quatorze complexes Hox suggérant une nouvelle duplication du génome spécifique de la famille des salmonidés[13].
Duplication d’une région chromosomique
[modifier | modifier le code]La duplication est parfois décrite comme une trisomie partielle. Il existe 2 types de duplication :
- duplication directe (ou en tandem) : le fragment se duplique dans le même sens ;
- duplication inverse (ou en miroir) : le fragment se duplique en sens inverse.
On peut également distinguer les duplications :
- intrachromosomique : le fragment est dupliqué sur le même chromosome à la suite d'un mécanisme de réparation non homologue de l'ADN ;
- interchromosomique : le fragment est dupliqué sur un autre chromosome à la suite d'une translocation chromosomique.
De nombreux syndromes résultent de la duplication d'un segment de chromosome, tel que le syndrome de Beckwith-Wiedemann ou la maladie de Charcot-Marie-Tooth type 1. Bien que souvent délétère pour un organisme, l'analyse des génomes de nombreuses espèces, et notamment le génome humain, a révélé la présence de nombreuses régions dupliquées.
Exemples de segments de chromosomes dupliqués dans le génome humain
[modifier | modifier le code]Le séquençage et l'analyse du génome humain ont révélé que de grosses portions de chromosomes étaient similaires[14]. 400 segments de chromosomes pendant l'évolution des primates ont subi de nombreux évènements de duplication intrachromosomiques (entre chromosomes identiques) et interchromosomiques (entre chromosomes différents) dont certaines, qui représentent 5 % du génome, sont spécifiques du génome humain. Ces segments dupliqués de chromosome sont localisés préférentiellement près des télomères et des centromères. Par ailleurs, ces régions dupliquées sont sensibles aux réarrangements chromosomiques et sont le site de cassures de translocations, délétions ou inversions[15]. Cependant, bien que délétères, les fragments de chromosomes dupliqués sont proportionnellement transcriptionnellement plus actifs que les régions uniques des génomes de primate. De plus, ces duplications répétées ont créé de nouvelles familles géniques uniques chez les hominoïdes[16].
Le Chromosome 15
[modifier | modifier le code]8,8 % du chromosome 15 correspond à des segments de chromosomes dupliqués dont 50 % sont des duplications intrachromosomiques[17]. Par exemple, dans la région 15q, une séquence « cœur » de 2920 paires de base a été identifiée. Elle est répétée 37 fois sur le chromosome 15, 2 fois sur le chromosome Y et une fois sur le chromosome 2 et 10. L'analyse des différentes séquences de cette portion de chromosome « cœur » a permis de récapituler l'histoire de cette séquence puisque le génome du chien et de la souris ne contiennent qu'une seule copie de cette séquence « cœur » sur le chromosome correspondant au chromosome 2 humain. Ainsi, cette séquence a été copiée par rétrotransposition sur le chromosome 10, puis un fragment plus important de 15 Kilobases a été copié sur le chromosome 15. Ce segment d'ADN de 15 Kb s'est ensuite dupliqué plusieurs fois sur le chromosome 15. Les deux copies de la séquence « cœur » initiale sur le chromosome Y est le résultat de la copie d'une séquence de 40 kb originaire du chromosome 15. Comme mentionné précédemment, les régions dupliquées sont des portions de chromosome fragiles et sensibles aux réarrangements chromosomiques. Les délétions chromosomiques dans la région dupliquée 15q11-q13 sont la cause des syndromes Prader-Willi et Angelman.
Syndrome Beckwith-Wiedemann
[modifier | modifier le code]Le syndrome de Beckwith-Wiedemann est caractérisé par une grande taille, une macroglossie et une organomégalie, et résulte dans un grand nombre de cas d'une duplication de la région terminale du chromosome 11 paternel. Cette région chromosomique contient deux gènes essentiels à la régulation de la croissance; les gènes IGF2 et H19. IGF2 est un facteur de croissance stimulant la prolifération cellulaire et H19 code un ARN non codant. L'expression de ces gènes est extrêmement régulée par un mécanisme complexe d'empreinte parentale. Sur le chromosome maternel, le gène IGF2 est réprimé et H19 est exprimé alors que sur le chromosome paternel, le gène IGF2 est exprimé et H19 réprimé. Le syndrome résulte d'une duplication partielle de la partie terminale du chromosome 11 paternel. Cette duplication apporte une copie supplémentaire du gène IGF2 sur le chromosome 11 conduisant à une augmentation de la production du facteur de croissance IGF2.
Duplication d'un gène
[modifier | modifier le code]Mécanismes de duplication génique
[modifier | modifier le code]Un gène particulier peut être dupliqué au cours de l'évolution. Il existe quatre principaux mécanismes permettant la duplication d'un gène ou de quelques gènes :
- par rétrotransposition : lors de la rétrotransposition d'un élément transposable, il peut y avoir transcription d'une partie de la région chromosomique à proximité de l'élément transposable, comprenant éventuellement un ou plusieurs gènes. Lorsque l'ARN est rétrotranscrit au sein du génome, le ou les gènes transcrits par accident sont alors copiés dans une autre région du génome ;
- par crossing-over inégal (lors de la méiose, les chromosomes homologues s'apparient sur les régions semblables, et peuvent éventuellement s'échanger). Ainsi, une partie de l'ADN de l'un des deux chromosomes homologues est transférée sur l'autre. On a ainsi une perte de gènes sur un chromosome et une duplication de gènes sur l'autre si cette portion de chromosome porte un ou plusieurs gènes. On obtient alors une disposition des gènes dupliqués les uns à la suite des autres, en tandem ;
- par échange ectopique : lors d'une cassure double brin, il peut y avoir recombinaison avec un site non homologue, puis élongation de la molécule d'ADN suivant la matrice d'ADN recombinante, recopiant ainsi une portion du génome après la cassure. La réparation de la cassure a ensuite pour conséquence l'intégration de cette région dupliquée au génome ;
- par un mécanisme de transfert horizontal de gènes : L'ADN exogène provient d'un organisme transducteur (virus, parasite, cellule morte, endosymbionte (mitochondrie, chloroplaste)). Cependant, ce mécanisme d'échange de matériel génétique génère rarement une duplication génique.
Importance des duplications géniques
[modifier | modifier le code]Les duplications de gènes sont des évènements extrêmement fréquents. Ainsi, le nombre de répétitions d'un même gène peut varier entre les individus de la même espèce, formant ainsi un polymorphisme du nombre de répétitions. L'augmentation du nombre de gènes est cependant contrebalancée par une perte de gènes elle aussi assez forte, principalement par dérive génétique, mais aussi par contre-sélection de l'augmentation de la concentration en protéines générée par l'augmentation du nombre de gènes codant cette protéine.
Conséquences de la duplication
[modifier | modifier le code]Il existe cinq scénario évolutifs faisant suite à une duplication génique :
- effet dose : Pour certains gènes l'augmentation du nombre de copies et donc de la quantité de protéines a un effet bénéfique. Par exemple, le gène codant la chimiokine CCL3L1 est situé dans une région fortement dupliquée et la variation du nombre de copies de CCL3L1 entre individus est un facteur de susceptibilité au développement d'un SIDA[18] ;
- adaptation cellulaire ou tissulaire : Les deux gènes gardent leur fonction originelle mais une des copies acquiert une expression spécifique d'un tissu (par exemple, l'expression du gène Glutamate déshydrogénase GLUD1 est ubiquitaire alors que la GLUD2 est exprimée spécifiquement dans le cerveau[19]) ou bien la protéine codée par un gène dupliqué a une localisation cellulaire différente (par exemple: la kinase Wee1 est nucléaire alors que la kinase Myt1 est localisée au niveau du réticulum endoplasmique) ;
- pseudogénisation : l'un des deux gènes dupliqués peut perdre sa fonction et ne plus être exprimé, se transformant ainsi en pseudogène. La pseudogénisation de nombreux gènes est impliquée dans la réponse immunitaire[Comment ?] ;
- subfonctionnalisation : si le gène originel avait deux "fonctions", l'un des deux gènes dupliqués peut perdre l'une des fonctions, et l'autre gène perdre la fonction complémentaire. Ainsi l'organisme est-il capable d'assurer les deux fonctions, mais elles sont alors assurées par deux protéines différentes. Cette subfonctionnalisation peut être sélectionnée, notamment si les deux fonctions présentent une certaine incompatibilité (expression dans des tissus différents, par exemple) ;
- néofonctionnalisation : comme la fonction sélectionnée sur le gène originel est codée par deux copies de gènes, la pression de sélection peut se relâcher sur l'une de ces copies. Il peut ainsi y avoir apparition d'innovations évolutives, et apparition d'une nouvelle fonction.
Modèle de Duplication-Dégénération: un mécanisme de fission génique
[modifier | modifier le code]Au cours de l'évolution des génomes, les gènes peuvent fusionner ou bien se fissurer. Une des conséquences de la subfonctionnalisation d'un gène dupliqué peut être la fission d'un gène en deux gènes différents. En effet, dans le cas où le gène originel code une protéine avec deux domaines fonctionnels distincts notés I et II, l'un des gènes dupliqués accumule des mutations (dégénération) dans la région correspondant au domaine II alors que le second gène accumule des mutations dans la région correspondant au domaine I. Il en résulte un gène 1 codant une protéine avec le domaine I et un second gène codant une protéine avec le domaine II. Ce mécanisme de fission génique a été mis en évidence chez la drosophile au cours de l'étude du gène Monkey-king[20].
Modèle de Bateson-Dobzhansky-Muller : Duplication du génome et incompatibilité génique
[modifier | modifier le code]L'isolement reproductif est un processus essentiel à la spéciation car elle permet d'amorcer la divergence et le maintien d'espèces distinctes. Si deux espèces proches sont adaptées à des environnements différents, leurs hybrides seront moins bien adaptés à ces environnements : on parle de faiblesse hybride. Ce phénomène peut être expliqué par le modèle Dobzhansky-Muller d'incompatibilité génique développé par Theodosius Dobzhansky en 1936 et Herman Joseph Müller en 1942. La redécouverte des travaux de William Bateson a amené certains auteurs à parler de modèle Bateson-Dobzhansky-Muller[21]. Le modèle propose que les interactions épistatiques négatives entre deux locus conduit à la faiblesse hybride. En 2000, Michael Lynch et Allan G. Force ont adapté le modèle d'incompatibilité génique à la duplication génique[22]. Ils proposèrent qu'à la suite d'une duplication génique, la perte génique réciproque est un facteur entrainant l'isolement reproductif. Ainsi, ce modèle expliquerait la corrélation entre des évènements de duplication complète du génome et la radiation évolutive de certains phyla. Par exemple, il a été démontré que chez l'ancêtre de la levure de boulanger, qui a subi une duplication complète du génome, la perte rapide des gènes dupliqués a favorisé l'émergence de nombreuses espèces de levures par un mécanisme d'incompatibilité génique[23].
Origine des microARN chez les plantes
[modifier | modifier le code]Les microARNs sont des ARN simple-brins de 21-23 nucléotides de long qui régulent l'expression génique post-transcriptionnellement. Chez la plante Arabidopsis thaliana, il existe une classe de microARNs qui présentent une forte homologie de séquence avec leurs gènes cibles et apparurent récemment au cours de l'évolution. Ces microARNs sont le résultat de duplications géniques inversées.
Duplication en tandem d'exons
[modifier | modifier le code]
La duplication en tandem d'exons correspond à une duplication interne d'un exon au sein du même gène. Ce type de duplication est une source d'innovation importante car il permet de générer au sein d'une même protéine de nouvelles fs. Une analyse complète des génomes d'Homo sapiens, de Drosophila melanogaster et du ver Caenorhabditis elegans ont montré 12291 cas de duplication en tandem d'exons. L'analyse des régions introniques a également mis en évidence 4660 exons dupliqués non caractérisés. Parmi ces exons potentiels, 35,1 % sont retrouvés dans les banques de données d'EST confirmant leur rôle potentiel[24].
Duplication d'une séquence nucléotique
[modifier | modifier le code]La réplication de ces paires de base est à l'origine de nombreuses maladies génétiques tels que le Syndrome de l'X fragile ou bien la Chorée de Huntington. Chez la bactérie Streptococcus pneumoniae, la duplication de 18 paires de base dans le gène rplV qui code la protéine ribosomique L22 est responsable de la résistance au macrolide. Cette duplication génère une duplication de 6 acides aminés à proximité du site d'interaction de la macrolide sur la sous-unité 23S du ribosome bloquant ainsi l'interaction de l'antibiotique avec le ribosome[25].
Duplication génique chez les procaryotes
[modifier | modifier le code]L'analyse des génomes procaryotes a révélé de nombreux gènes paralogues. Suivant les espèces bactériennes, la proportion de gènes paralogues varie de 7 % du génome chez Rickettsia conorii à 41 % chez Streptomyces coelicolor. Sur l'ensemble des 106 génomes bactériens séquencés en 2004, en moyenne 25 % du génome bactérien est constitué de gènes paralogues. Il existe également une corrélation très forte entre la taille du génome bactérien et la proportion de gènes dupliqués[26].
Mécanismes
[modifier | modifier le code]Il existe trois mécanismes de duplication génique chez les procaryotes :
- au cours de la réplication de l'ADN à la suite d'un mécanisme de recombinaison homologue favorisé par la présence de séquences répétées tel que les opérons des ARN ribosomiques, les transposons et les séquences d'insertion IS ;
- à la suite d'une cassure double brin de l'ADN, un gène peut être dupliqué par réplication de type rolling circle ;
- un transfert de gène horizontal entre espèces bactériennes. Les deux copies homologues sont appelées xénologues.
Chez Escherichia coli K12, 16 % des gènes homologues sont xénologues suggérant que le transfert horizontal de gène a un faible impact sur la proportion de gènes paralogues. En moyenne, 15 % des gènes paralogues sont organisés en tandem suggérant des évènements de duplication génique en tandem qui peuvent résulter en la duplication d'opéron.
Chez les procaryotes, aucune duplication ancestrale complète du génome n'a été mise en évidence. Cependant, certaines bactéries sont polyploïdes au cours d'un stade de développement telles que Buchnera aphidicola dont le nombre de copies du génome varie en fonction du stade de développement de son hôte, le puceron vert du bois[27].
Rôle au cours de l'évolution
[modifier | modifier le code]
La plupart des génomes eucaryotes portent un nombre important de gènes dupliqués fonctionnels dont une grande majorité serait apparue il y a 10 à 100 millions d'années[28]. À la suite d'événements massifs de duplication, de l'ordre de 20 à 50 % des gènes dupliqués sont conservés. Cette conservation suggère l'existence d'un mécanisme de sélection naturelle qui compenserait la production de pseudogènes. Après une duplication, il peut y avoir une séparation des fonctions ancestrales entre les deux copies, par exemple dans des tissus différents (phénomène dit de sous-fonctionnalisation) ; une apparition d'une nouvelle fonction pour une des copies (phénomène dit de néofonctionnalisation) ; chaque copie peut connaître une perte ou une réduction de l'expression de ses fonctions par des mutations dégénératives[29].
L'évolution du génome bactérien est profondément influencée par des échanges géniques entre différentes espèces bactériennes. En effet, l'évolution des voies métaboliques chez E.coli résulte principalement d'échanges géniques horizontaux[30].Cependant, la duplication génique semble également contribuer à l'évolution bactérienne. Les gènes préférentiellement dupliqués sont impliqués dans le métabolisme des acides aminés, des ions inorganiques et la régulation transcriptionnelle. Chez certaines espèces, l'expansion génique d'une catégorie de gènes a été proposée comme étant une adaptation évolutive. Par exemple, la complexité de la paroi bactérienne chez les mycobactéries peut être expliquée par une duplication spécifique à ce genre bactérien des gènes impliqués dans le métabolisme et le transport des lipides.
Amplification génique et résistance aux antibiotiques
[modifier | modifier le code]
Les bactéries peuvent s'adapter à la toxicité d'un antibiotique grâce à une grande batterie de mécanismes résultant soit de mutations ponctuelles ou bien d'un transfert horizontal de gènes. Les mécanismes principaux de résistance sont :
- la dégradation de l'antibiotique ;
- la séquestration de l'antibiotique ;
- la prévention de l'absorption de l'antibiotique ;
- l'expulsion de l'antibiotique hors de la bactérie ;
- bloquer l'interaction entre l'antibiotique et la molécule cible.
L'adaptation d'une bactérie à un antibiotique s'effectue en deux étapes :
- acquisition de la résistance qui dans la plupart des cas résulte en une diminution de la valeur sélective (fitness) de la bactérie ;
- adaptation de la bactérie à la résistance par l'accumulation de mutations/modifications compensatoires qui dans de nombreux cas sont le résultat d'amplification génique soit du gène conférant la résistance ou bien d'un autre gène.
Les amplifications géniques sont instables et peuvent être perdues en absence de pression de sélection par recombinaison homologue. Ainsi, les gènes dupliqués peuvent être maintenus dans des souches bactériennes où la protéine Rec A est déficiente. Cependant, il existe des mécanismes de perte de gènes dupliqués indépendants de Rec A qui sont peu connus[31].
Amplification génique sur un plasmide
[modifier | modifier le code]Au début des années 1970, la découverte des plasmides R porteurs de gènes de résistances aux antibiotiques a permis de mettre en évidence pour la première fois le rôle de la duplication génique dans le phénomène de résistance aux antibiotiques. Le plasmide NR1 augmente en taille chez Proteus mirabilis en présence de faible concentration de chloramphénicol, streptomycine et sulfonamide. La région amplifiée du plasmide est appelée déterminant-r (r pour résistance) et est flanquée par des séquences répétées directes IS1 (Insertion Sequence 1) qui permettent l'amplification de la région grâce aux homologies de séquence entre les séquences IS1 entrainant une recombinaison homologue entre chromatides sœurs[32],[33]. Dans de nombreuses bactéries, la résistance associée à l'amplification génique sur un plasmide est souvent associée à la présence de séquence IS. Cependant, il existe quelques cas où des régions homologues imparfaites peuvent permettre l'amplification de gènes de résistances tel que l'amplification du gène tetL sur le plasmide pAMα1 qui confère la résistance à la tétracycline chez Enterococcus faecalis[34].
Amplification génique sur le chromosome bactérien
[modifier | modifier le code]L'amplification des gènes chromosomique est similaire à celle observée sur les plasmides bactériens. Par exemple, il a été observé une amplification de la β-lactamase chez Escherichia coli et Yersinia enterocolitica conférant une résistance aux β-lactamines par des mécanismes dépendants et indépendants de la protéine Rec A[35]. Cependant, il existe des exemples particuliers où seulement quelques paires de base sont dupliquées. Chez Staphylococcus aureus et Yersinia enterocolitica, la duplication du site d'entrée du ribosome du gène de résistance aux macrolides appelé ermA (pour erythromycin resistance methylase A) entraine une stimulation de la traduction de ermA[36]. Enfin chez la bactérie Streptococcus pneumoniae, la duplication de 18 paires de base dans le gène rplV qui code la protéine ribosomique L22 est responsable de la résistance aux macrolides. Cette duplication génère une duplication de 6 acides aminés à proximité du site d'interaction de la macrolide sur la sous-unité 23S du ribosome bloquant ainsi l'interaction de l'antibiotique avec le ribosome[25]. La plupart des amplifications géniques conférant des résistances aux antibiotiques ont été induites dans les laboratoires de recherche. Au cours d'essai clinique, l'identification d'amplification génique est assez rare probablement due à l'instabilité des amplifications géniques. Lors d'un criblage de souches de Streptococcus agalactiae, deux souches résistantes aux sulfonamides et au triméthoprime ont été isolées avec une amplification contenant l'opéron folCEPBK impliqué dans la synthèse de la vitamine B9[37].
Notes et références
[modifier | modifier le code]Notes
[modifier | modifier le code]- Cette duplication est présente chez tous les Vertébrés, mais absente chez les autres Chordés.
- Cette triplication est présente chez tous les Cyclostomes, mais absente chez les Gnathostomes.
- Cette seconde duplication est présente chez tous les Gnathostomes, mais absente chez les Cyclostomes.
Références
[modifier | modifier le code]- Wong AJ, Ruppert JM, Eggleston J, Hamilton SR, Baylin SB, Vogelstein B. Gene amplification of c-myc and N-myc in small cell carcinoma of the lungScience, 1986 Jul 25;233(4762):461-4
- Escot C, Theillet C, Lidereau R, Spyratos F, Champeme MH, Gest J, Callahan R. Genetic alteration of the c-myc protooncogene (MYC) in human primary breast carcinomas Proc Natl Acad Sci U S A, 1986 Jul;83(13):4834-8
- Kellis, M, Birren, BW & Lander, ES. Proof and evolutionary analysis of ancient genome duplication in the yeast Saccharomyces cerevisiae Nature 2004 428: 617-624.
- Jaillon, O. et al.; The grapevine genome sequence suggests ancestral hexaploidization in major angiosperm phyla Nature. 2007 Sep 27;449(7161):463-7
- Hervé Le Guyader, « Le double effet du génome de la myxine », Pour la science, no 559, , p. 92-94 (présentation en ligne, lire en ligne
 [PDF], consulté le ).
[PDF], consulté le ).
- Leitch, A.R. Leitch, I.J. Genome plasticity and the diversity of polyploid plants Science. 2008 Apr 25;320(5875):481-3
- Lim, K.Y. Soltis, D.E. et al. Rapid chromosome evolution in recently formed polyploids in Tragopogon (Asteraceae) PLoS One 2008;3(10):e3353. Epub 2008 Oct 9[1] .
- Ohno S (1970). Evolution by Gene Duplication. London: Allen and Unwin, (ISBN 0-04-575015-7).
- Makałowski W (2001). "Are we polyploids? A brief history of one hypothesis." Genome research 11(5): 667–670, , DOI 10.1101/gr.188801
- Dehal, P. Boore, J.L. (2005). "Two round of whole genome duplication in the ancestral vertebrate" PLoS Biology 3(10): e314
- Lynch, V.J. Wagner, G.P: (2009). "Multiple chromosomal rearrangements structured the ancestral vertebrate Hox-bearing protochromosomes" PLoS Genetics 5(1): e1000349
- Amores, A. Force A. et al. (1998). "Zebrafish Hox cluster and vertebrate genome evolution" Science Nov 27;282(5394):1711-4
- Moghadam HK, Ferguson MM, Danzmann RG. (2005)"Evolution of Hox clusters in Salmonidae: a comparative analysis between Atlantic salmon (Salmo salar) and rainbow trout (Oncorhynchus mykiss)" J Mol Evol. 2005 Nov;61(5):636-49
- Bailey, J.A. Eichler, E.E. Primate segmental duplications: crucibles of evolution, diversity and disease Nat Rev Genet. 2006 Jul;7(7):552-64
- Linardopoulou, E.V. Williams, E.M. Fan, Y. Friedman, C. Young, J.M. Trask, B.J. Human subtelomeres are hot spots of interchromosomal recombination and segmental duplication Nature. 2005 Sep 1;437(7055):94-100
- Zhiang, Z. et al. Ancestral reconstruction of segmental duplications reveals punctuated cores of human genome evolution Nature genetics, Volume 39, Number 11, pages 1361-68
- Zody M.C. et al. Analysis of the DNA sequence and duplication history of human chromosome 15 2006 Nature, Mar 30;440(7084):671-5
- Gonzalez E, Kulkarni H, Bolivar H, Mangano A, Sanchez R, Catano G, Nibbs RJ, Freedman BI, Quinones MP, Bamshad MJ, Murthy KK, Rovin BH, Bradley W, Clark RA, Anderson SA, O'connell RJ, Agan BK, Ahuja SS, Bologna R, Sen L, Dolan MJ, Ahuja SK. The influence of CCL3L1 gene-containing segmental duplications on HIV-1/AIDS susceptibility. Science 2005 Mar 4;307(5714):1434-40
- Burki F, Kaessmann H. Birth and adaptive evolution of a hominoid gene that supports high neurotransmitter flux. Nat Genet. 2004 Oct;36(10):1061-3. Epub 2004 Sep 19
- Wang,W. Yu, H. Long, M. Duplication-degeneration as a mechanism of gene fission and the origin of new gene in Drosophila species Nature Genetics vol36 (5) 52327
- Orr, HA. Dobzhansky, Bateson, and the genetics of speciation Genetics. 1996 Dec;144(4):1331-5.
- Lynch, M. Force AG. The origin of interspecies genomic incompatibility via gene duplication The american naturalist 137, 515-526 (1991)[2]
- Scannell DR, Byrne KP, Gordon JL, Wong S, Wolfe KH. Multiple rounds of speciation associated with reciprocal gene loss in polyploid yeasts Nature. 2006 Mar 16;440(7082):341-5Nature. 2006 Mar 16;440(7082):341-5.
- (en) Letunic I, Copley RR, Bork P, « Common exon duplication in animals and its role in alternative splicing », Hum Mol Genet., vol. 11, no 13, , p. 1561–7 (PMID 12045209, lire en ligne)
- Musher D, et al. Emergence of macrolide resistance duting treatment of pneumococcal pneumonia New England Journal of Medecine. 2002 346:630-631.
- Gevers D, Vandepoele K, Simillion C, Van der Peer Y, Gene duplication and biased functional retention of paralogs in bacterial genomes Trends in Microbiology. 2004 April;12(4):341-5.
- Komaki K, Ishikawa H, Genomic copy number of intracellular bacterial symbionts of Aphids varies in response to developmental stage and morph of their host Insect Biochem Mol Biol. 2000 30:253-8.
- (en) R. C. Cronn, R. L. Small & J. F. Wendel (1999). Duplicated genes evolve independently after polyploid formation in cotton. Proc Nat/Acad Sci USA 96(25): 14406-11.
- Thierry Lefevre, Michel Raymond, Frédéric Thomas, Biologie évolutive, De Boeck Superieur, , p. 252.
- Pal C, Papp B, Lercher MJ, "Adaptative evolution of bacterial metabolique networks by horizontal gene transfert" Nature Genetics, 2005 37:1372-1375
- Lovett ST, Gluckman TJ,Simon PJ, Sutera VA, Drapkin PT, "A sister-strand exchange mechanism for recA-independent deletion of repeated DNA sequences in Escherichia coli" Genetics, 1993 135:631-642
- Falkow S, Citarella RV, Wohlhieter JA. "The molecular nature of R-factors" Journal of Molecular Biology, 1966 17:102-116
- Perlman D, Rownd RH, "Transition of the R factor NR1 and Proteus mirabilis: molecular structure and replication of NR1 deoxyribonucleic acid" Journal of bacteriology, 1975 123:1013-1034
- Yagi Y, Clewell DB, "Plasmid-determined tetracycline resistance in streptococcus faecalis: tandemly repeated resistance determinantd in amplified forms of pAMα1" Journal of of Molecular Biology, 1976 102:503-600
- Seoane A, Sanchez E, Garcia-lobo JM, "Tandem amplification of a 28 kilobases region from Yersinia enterocolitica chromosome containing the blaA gene " Antimicrobial agents chemotherapy,2003 47:682-688
- Yoon EJ, Kwon AR, Min YH, Choi EC, "Foggy D-shaped zone of the inhibition in Staphylococcus aureus owing to a dual character of both inducible and constitutive resistance to macrolide-lincosamide-streptagramin B" Journal of Antimicrobial chemotherapy,2008 61:533-540
- Brochet M, Couve E, Zouine M, Poyert C, Glaser P, "A naturally occuring gene amplification leading to sulfonamide and trimethoprim resistance in Streptococcus agalactiae " Journal of bacteriology, 2008 190:672-680
